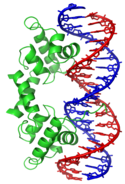
Transkriptionsfaktor
Transkriptionsfaktorer är proteiner som på något sätt, direkt eller indirekt, är involverade i transkriptionen av gener. Transkriptionen är en del av genetikens centrala dogm och kan förenklat sägas vara processen där en del av cellens DNA (en gen) kopieras till en RNA-kopia (mRNA) som sedan kan användas som ritning för syntesen av ett protein. Transkriptionsfaktorer är nödvändiga för att en gen skall kunna uttryckas och för att styra hur högt uttryck den skall ha (hur många mRNA-kopior den skall ge upphov till).
Alla transkriptionsfaktorer kännetecknas av att de har en eller flera regioner som binder till särskilda DNA-sekvenser, vilket innebär att vissa transkriptionsfaktorer är mycket specifika i vilka gener de reglerar och andra är väldigt generella. Alla transkriptionsfaktorer innehåller dock DNA-bindande regioner vilket särskiljer dem från andra proteiner som är involverade i genuttryck såsom t.ex. kromatinremodellerare och metylaser som ej har DNA-bindande regioner[1].
Allmänt
Väldigt tidigt i genomikens tidevarv upptäcktes att den uppfattade komplexiteten hos en art inte korrelerade särskilt bra till mängden arvsmassa eller ens antal gener. Människans genom består t.ex. av ca. 20 000 gener fördelade på ett genom som är ca. 3,2 Gb stort [2] medan t.ex. granens (Picea abies) genom består av c.a. 28 000 gener fördelade över 20 Gb arvsmassa[3]. Samtidigt får potatisen (Solanum tuberosum) nöja sig med ca. 0,85 Gb som innehåller ca. 39 000 gener[4]. Vad vi vet idag är att många gener återfinns i många olika släkten och att de stora skillnaderna i genomstorlek och antal gener till stor del beror på genduplikationer, alltså att olika arter har olika många kopior av samma gener[5]. Skillnaden i antalet unika gener är betydligt mindre och innebär att vertebrater generellt endast innehar dubbelt så många unika gener som invertebrater, trots deras stora skillnader i komplexitet[5]. Svaret ligger till stor del i att mer komplexa organismer har utvecklat mer komplexa nätverk av transkriptionsfaktorer som tillsammans reglerar stora, och mycket specifika, grupper av gener.
En liknelse som går att använda är att transkriptionsfaktorerna agerar dirigenter för övriga geners aktivitet likt en dirigent för en orkester som dirigerar de enskilda musikerna i orkestern. Genom att byta ut dirigenten erhålls ett annat musikaliskt intryck än genom den tidigare trots att i stort sett samma musiker är aktiva. På ett liknande sätt kan små förändringar i aktiva transkriptionsfaktorer påverka mycket stora grupper av gener vars sammanvägda resultat blir att cellerna utvecklas till olika typer av celler t.ex. muskelceller och nervceller trots att de i stort byggs av samma gener.
Transkriptionsfaktorer delas in i generella eller basala transkriptionsfaktorer som är transkriptionsfaktorer som alltid krävs för transkriptionsinitiering och regulatoriska eller specifika transkriptionsfaktorer som styr nivån av aktivitet hos genen (hur högt uttryckt genen är).
Generella transkriptionsfaktorer
Generella transkriptionsfaktorer (GTF) är transkriptionsfaktorer som alltid behövs för att transkriptionen av en gen skall kunna initieras. GTF binder i allmänhet i direkt anslutning till genen som skall transkriberas i dess promotorregion[6]. Till dessa hör bland annat TFIIA och TFIIB, där TFII refererar till att de interagerar med RNA-polymeras II, som syntetiserar mRNA i eukaryota celler.
Generella transkriptionsfaktorernas roll i transkriptionsinitieringen hos eukaryoter:
Transkription med RNA-polymeras II initieras genom att transkriptionsfaktorn TFIID binder till en DNA-sekvens på promotorn som kallas TATA-box, som befinner sig omkring 25 baspar uppströms från genen som ska transkriberas och består av en sekvens av adenin och tymin, oftast i basparsföljden TATAAA.
Inbindningen orsakar en förändring i DNA-strukturen som gör att fler transkriptionsfaktorer samt RNA-polymeras II kan bilda ett komplex vid TATA-boxen.
En annan generell transkriptionsfaktor, TFIIH, kommer med hjälp av ATP-hydrolys att tvinga isär DNA-dubbelhelixen så att RNA-polymeras kan binda till DNA-strängen och påbörja transkriptionen.
Transkriptionen initieras då TFIIH fosforylerar RNA-polymeras II. Detta medför att dess konformation förändras vilket gör att RNA-polymeraset kan dissociera från komplexet men fortfarande vara bunden till DNA-strängen, så att hela genen kan transkriberas. Fosforyleringen bidrar även till att proteiner som ska processa RNA efter transkriptionen kan binda in till polymeraset.
Regulatoriska transkriptionsfaktorer
De regulatoriska, eller specifika transkriptionsfaktorerna varierar från gen till gen, till skillnad från de generella transkriptionsfaktorerna som i allmänhet är lika mellan olika gener. Regulatoriska transkriptionsfaktorer binder oftast till DNA:t längre uppströms från den gen i vars reglering de är involverade i. Studier har visat på att transkriptionsfaktorer även kan binda nedströms transkriptionens startplats[7] men det är i dag ännu obevisat att denna bindning är specifikt involverad i transkriptionsinitieringen som sker uppströms.
De regulatoriska transkriptionsfaktorerna kan antingen inhibera (hämma) transkription och kallas då repressorer eller stimulera transkription (aktivera) och kallas då aktivatorer. Förenklat kan man säga att alla transkriptionsfaktorer och transkriptionskofaktorer bildar ett initieringskomplex där effekten de olika repressorerna och aktivatorerna vägs samman och avgör hur frekvent initiering av transkriptionen skall ske och därmed hur aktiv genen skall vara. Observera dock att det finns fler nivåer av kontroll av genuttryck ovanför transkriptionen.
Struktur
Transkriptionsfaktorer består av ett antal olika regioner där en eller flera DNA-bindande regioner samt en eller flera transaktiveringsregioner eller en eller flera transrepressionsregioner återfinns hos alla transkriptionsfaktorer[1].
DNA-bindande regioner
DNA-bindande regioner är proteinstrukturer som känner igen och binder till DNA, antingen genom sekvensspecifik bindning eller icke-sekvensspecifik bindning[1]. Interaktion med DNA:t kan ske genom minor eller major groove där sekvensspecifik bindning underlättas via direktinteraktion med baserna, eller via DNA:ts socker-fosfatryggrad som generellt innebär icke-sekvensspecifik bindning. DNA-sekvenserna som de DNA-bindande motiven binder till kallas för DNA-motiv och består i allmänhet av en eller flera sekvenser som består av 6-10 nukleotider vardera[8]. Nedan presenteras några av de vanligaste förekommande DNA-bindande regioner som har blivit identifierade. Observera att listan inte på något sätt är fullständig.

DNA-bindande region | Beskrivning | PFAM |
Helix-turn-helix | Helix-turn-helix är en ca. 20 aminosyror lång DNA-bindande region som består av två α-helixar, en i C-terminalen och en i N-terminalen av den DNA-bindande regionen. I de flesta fall bidrar en av α-helixarna mer till DNA-bindningen än den andra och kallas då för igenkänningshelix (recognition helix)[9]. De är vanligt förekommande i repressorer. Helix-turn-helix-strukturer kan delas in i åtminstone fem olika klasser baserat på deras sekundär- och tertiärstruktur[10]. | Varierar med klass. |
Zinkfinger (zinc finger) | Zinkfingrar är DNA-bindande regioner (även om det finns zinkfingrar som t.ex. binder andra proteiner) som är mycket varierande i form och funktion. Namnet kommer ifrån att de tidigaste identifierade zinkfingrarna bestod av korta α-helixar som stack ut från proteinet likt fingrar och band till en zink-kofaktor[11][12]. Sedan dess har minst fem olika klasser identifierats vars struktur varierar kraftigt[13]. Vissa zinkfingrar nyttjar sig av andra kofaktorer än zink såsom järn medan vissa helt saknar metall-kofaktorer[13]. Tillika varierar inbindningen kraftfullt men i allmänhet binder zinkfingrar till DNA:ts major groove. | Varierar med klass. |
Basic helix-loop-helix | Basic helix-loop-helix består av två ALPHA-helixar som binds samman med en aminosyrekedja i loop-form. I allmänhet är en av helixarna mindre och binder till en annan α-helix medan den större står för den sekvensspecifika DNA-bindningen[14]. Basic helix-loop-helix regioner binder i allmänhet till en så kallad E-box som har konsensussekvensen CANNTG[14]. | PF00010 |
Basic leucin zipper | Basic leucin zipper-regionen består i allmänhet av 60-80 aminosyror som bildar två subenheter som vardera består av två α-helixar som bildar dels en evolutionärt mycket bevarad region som allmänt binder till DNA och en betydligt mer varierande region med sekvensspecifik bindning[15][16]. Namnet leucin zipper kommer ifrån att den sekvensspecifika regionens α-helixar nyttjar utstickande leuciner (samt i mindre omfattning även argininer) för att interagera med DNA-sekvensen[15][16]. Detta åstadkoms genom att leuciner (eller argininer) förekommer som var sjunde aminosyra i aminosyrasekvensen som då kan liknas vid en regelbunden dragkedja[16]. | PF00170 |
HMG-box | HMG-boxar består av tre stycken α-helixar som binds samman med loopar[17]. HMG-boxar binder enbart till DNA med annan struktur än den allmänna B-strukturen och har därför hög affinitet till DNA som påverkas av flera DNA-beroende processer såsom transkription och replikering[18]. Många protein med HMG-boxar är därför involverade i dessa processer. | PF00505 |
B3-region | B3-regionen är en DNA-bindande region som enbart förekommer hos högre växter. Den består av ca. 100-120 aminosyror som genom sju β-flak och två α-helixar bildar en pseudotunna som kan interagera med DNA:ts major groove[19]. | PF02362 |
Homeobox | Homeobox DNA-bindande regioner utgör en viktig grupp av DNA-bindande proteiner hos eukaryoter som är involverade i organismers utveckling[20]. En homeobox är ca. 120 aminosyror lång och deras DNA-bindande region består av en helix-turn-helix struktur där den tredje α-helixen binder till DNA:ts major groove[21]. | PF00046 |
Utöver dessa så finns ytterligare flera DNA-bindande regioner som är vanligt förekommande i transkriptionsfaktorer såsom Wor3-regionen[21] eller OB-fold regioner[22]. Då viss förvirring råder kring vilka DNA-bindande regioner som klassificeras som klasser av större grupper så kan man ibland stöta på t.ex. i fallet med winged helix som ibland beskrivs som en egen grupp men snarare är en klass av helix-turn-helix-gruppen[23].
Transaktiverings- och transrepressionsregioner
Transaktiveringsregioner är regioner hos transkriptionsfaktorn som, när aktiva, binder till andra proteiner som är viktiga för transkriptionen av genen. Transaktiveringsregionen kan dels binda direkt till transkriptionsmaskineriet och på så sätt initiera transkriptionen eller vi så kallade transkriptionskofaktorer som agerar länk mellan transkriptionsfaktorn och transkriptionsmaskineriet. Transrepressionsregioner är liknande regioner men istället för att initiera eller binda proteiner som initierar transkription (så kallade transkriptionkoaktivatorer) så inhiberar eller binder regionen protein (så kallade transkriptionskorepressorer) som inhiberar transkriptionsinitieringen.
Signal sensing domain
Signal sensing domain (ungefär signaligenkännande region) är en region hos vissa transkriptionsfaktorer som har möjligheten att binda en ligand, t.ex. ett näringsämne eller ett hormon, eller fosforyleras och då aktiveras eller inhiberas[24]. T.ex. kan ett hormon binda till transkriptionsfaktorn som då genom att ändra sin struktur blottar sin transaktiveringsregion varpå transkriptionskoaktivatorer kan binda och initiera transkriptionen. På så sätt kan genen stängas av och sättas på mycket snabbt via t.ex. hormoner. Signal sensing domain måste inte vara placerad på transkriptionsfaktorn utan kan vara belägen på en separat transkriptionskofaktor.
Cis-sekvenser
Cis-sekvenser, även kallade regulatoriska sekvenser, är regioner uppströms den gen som transkriptionsfaktorerna påverkar. Termen regulatoriska sekvenser är dock inte unik för sekvenser som influerar transkriptionsfaktorers funktion utan är mer generell och omfattar alla sekvenser involverade i regleringen av genuttryck. Cis-sekvenser delas ofta upp i promotorregioner, silencerregioner, enhancerregioner och insulatorregioner. Promotorer är cis-sekvenser direkt uppströms från genens transkriptionsstartsekvens. Promotorns konstruktion skiljer sig mellan eukaryoter och prokaryoter där de eukaryota promotorerna är betydligt mer komplexa an prokaryoternas dito. Enhancerregioner är regioner som kan ligga avsevärt uppströms promotorn och har förmågan att binda aktivatorer och via att DNA-strängen är flexibel interagera med transkriptionsfaktorerna i promotorregionen. Silencerregionen är motsvarande regioner men binder repressorer.
En sekvens som specifikt binder en eller flera enskilda transkriptionsfaktorer kallas för responselement. Dessa består i allmänhet av en DNA-sekvens 6-10 baspar lång[6].
Klassificering av transkriptionsfaktorer
Transkriptionsfaktorer klassificeras i allmänhet efter en av tre olika faktorer; (i) mekanism, (ii) funktion eller (iii) likheter i aminsoyrasekvensen.
Mekanism
Det finns två mekanistiska klasser av transkriptionsfaktorer.
- Generella transkriptionsfaktorer – transkriptionsfaktorer som interagerar med core promotor region hos alla klass-2-gener
- Uppströmstranskriptionsfaktorer – transkriptionsfaktorer ej tillhörande de generella transkriptionsfaktorerna. Begreppet mycket snarlikt regulatoriska transkriptionsfaktorer.
Funktion
Transkriptionsfaktorerna klassificeras beroende på deras funktion[25]:
I. Constitutively active – Alltid närvarande i samtliga celler (generella transkriptionsfaktorer)
II. Conditionally active – Kräver någon form av aktivering
- II.A Developmental – Cellspecifika. Starkt reglerade initial men när de väl aktiverats fortsätter de i allmänhet vara aktiva.
- II.B Signal-dependent – Kräver en yttre signal för aktivering.
- II.B.1 Extracellular ligand-dependent – Kräver aktivering av ligander som ej kan syntetiseras av cellen själv.
- II.B.2 Intracellular ligand-dependent – Kräver aktivering av ligander som kan syntetiseras av cellen.
- II.B.3 Cell membrane receptor-dependent – Aktivering beroende av second messenger fosforylering.
- II.B.3.a Resident nuclear factors – Befinner sig i cellkärnan oavsett aktiveringsstatus.
- II.B.3.b Latent cytoplasmic factors – Aktiv form transporteras in i cellkärnan från cytosolen.
Sekvenslikheter
Följande klassificering bygger på sekvenslikheter och aktiva element[26][27]
1 Superklass: Basic Domains
- 1.1 Klass: Leucine zipper transkriptionsfaktorer (bZIP)
- 1.1.1 Familj: AP-1-liknande komponenter
- 1.1.2 Familj: CREB
- 1.1.3 Familj: C/EBP-liknande transkriptionsfaktorer
- 1.1.4 Familj: bZIP / PAR
- 1.1.5 Familj: Plant G-boxbindande transkriptionsfaktorer
- 1.1.6 Familj: ZIP enbart
- 1.2 Klass: Helix-loop-helix transkriptionsfaktorer (bHLH)
- 1.2.1 Familj: Ubiquitous (Klass A) transkriptionsfaktorer
- 1.2.2 Familj: Myogenic transkriptionsfaktorer (MyoD)
- 1.2.3 Familj: Achaete-Scute
- 1.2.4 Familj: Tal/Twist/Atonal/Hen
- 1.3 Klass: Helix-loop-helix / leucine zipper transkriptionsfaktorer (bHLH-ZIP)
- 1.3.1 Familj: Ubiquitous bHLH-ZIP transkriptionsfaktorer;
- 1.3.2 Familj: Cell-cycle kontrollerande transkriptionsfaktorer
- 1.4 Klass: NF-1
- 1.4.1 Familj: NF-1 (A, B, C, X)
- 1.5 Klass: RF-X
- 1.5.1 Familj: RF-X (1, 2, 3, 4, 5, ANK)
- 1.6 Klass: bHSH
2 Superklass: Zinc-innehållande DNA-bindande regioner
- 2.1 Klass: Cys4 zinkfinger av kärnreceptortyp
- 2.1.1 Familj: Steroidhormonreceptorer
- 2.1.2 Familj: Tyreoideahormonreceptorliknande transkriptionsfaktorer
- 2.2 Klass: skiftande Cys4 zinkfingrar
- 2.2.1 Familj: GATA-transkriptionsfaktorer
- 2.3 Klass: Cys2His2 zinkfingerregioner
- 2.3.1 Familj: Ubiquitous-transkriptionsfaktorer, inkluderar TFIIIA, Sp1
- 2.3.2 Familj: Utvecklings-/cellcykelreglerande; inkluderar Krüppel
- 2.3.4 Familj: Stora transkriptionsfaktorer med NF-6B-liknande bindningsegenskaper
- 2.4 Klass: Cys6 cystein-zink-kluster
- 2.5 Klass: Zinkfingrar med varierande beståndsdelar
3 Superklass: Helix-turn-helix
- 3.1 Klass: Homeoregion
- 3.1.1 Familj: Homeoregion enbart; inkluderar Ubx
- 3.1.2 Familj: POU-region transkriptionsfaktorer; inkluderar Oct
- 3.1.3 Familj: Homeoregion med LIM-region
- 3.1.4 Familj: Homeoregion samt zinkfinger-regioner
- 3.2 Klass: Paired box
- 3.2.1 Familj: Paired samt homeo domain
- 3.2.2 Familj: Paired domain endast
- 3.3 Klass: Fork head / winged helix
- 3.3.1 Familj: Utvecklingsregulatorer; inkluderar forkhead
- 3.3.2 Familj: Vävnadsspecifika regulatorer
- 3.3.3 Familj: Cellcykelkontrollerande transkriptionsfaktorer
- 3.3.0 Familj: Andra regulatorer
- 3.4 Klass: Heat Shock-transkriptionsfaktorer
- 3.4.1 Familj: HSF
- 3.5 Klass: Tryptofankluster
- 3.5.1 Familj: Myb
- 3.5.2 Familj: Ets-typ
- 3.5.3 Familj: Interferon reglerande faktorer
- 3.6 Klass: TEA (transcriptional enhancer factor)-region
- 3.6.1 Familj: TEA (TEAD1, TEAD2, TEAD3, TEAD4)
4 Superklass: beta-Scaffold-transkriptionsfaktorer med minor groove-interaktion
- 4.1 Klass: RHR
- 4.1.1 Familj: Rel/ankyrin; NF-kappaB
- 4.1.2 Familj: Ankyrin enbart
- 4.1.3 Familj: NFAT (Nuclear Factor of Activated T-cells) (NFATC1, NFATC2, NFATC3)
- 4.2 Klass: STAT
- 4.2.1 Familj: STAT
- 4.3 Klass: p53
- 4.3.1 Familj: p53
- 4.4 Klass: MADS box
- 4.4.1 Familj: Reglerare av differentiering; inkluderar (Mef2)
- 4.4.2 Familj: Responders to external signals, SRF (serum response factor) (SRF)
- 4.4.3 Familj: Reglerare av metabolism (ARG80)
- 4.5 Klass: beta-Barrel alpha-helix transkriptionsfaktorer
- 4.6 Klass: TATA-bindande protein
- 4.6.1 Familj: TBP
- 4.7 Klass: HMG-box
- 4.7.1 Familj: SOX-gener, SRY
- 4.7.2 Familj: TCF-1 (TCF1)
- 4.7.3 Familj: HMG2-related, SSRP1
- 4.7.4 Familj: UBF
- 4.7.5 Familj: MATA
- 4.8 Klass: Heteromeric CCAAT-transkriptionsfaktorer
- 4.8.1 Familj: Heteromeric CCAAT-transkriptionsfaktorer
- 4.9 Klass: Grainyhead
- 4.9.1 Familj: Grainyhead
- 4.10 Klass: Cold-shock-regioninnehållande transkriptionsfaktorer
- 4.10.1 Familj: csd
- 4.11 Klass: Runt
- 4.11.1 Familj: Runt
0 Superklass: Övriga transkriptionsfaktorer
- 0.1 Klass: Copper fist-protein
- 0.2 Klass: HMGI(Y) (HMGA1)
- 0.2.1 Familj: HMGI(Y)
- 0.3 Klass: Pocket domain
- 0.4 Klass: E1A-liknande transkriptionsfaktorer
- 0.5 Klass: AP2/EREBP-relaterade transkriptionsfaktorer
- 0.5.1 Familj: AP2
- 0.5.2 Familj: EREBP
- 0.5.3 SuperFamilj: AP2/B3
- 0.5.3.1 Familj: ARF
- 0.5.3.2 Familj: ABI
- 0.5.3.3 Familj: RAV
Referenser
Noter
- ^ [a b c] Latchman, David S. (1997). ”Transcription factors: An overview”. The International Journal of Biochemistry & Cell Biology 29 (12): sid. 1305-1312. doi:. http://www.sciencedirect.com/science/article/pii/S135727259700085X.
- ^ Consortium, International Human Genome Sequencing. ”Finishing the euchromatic sequence of the human genome” (på engelska). Nature 431 (7011): sid. 931-945. doi:. ISSN 0028-0836. http://www.nature.com/nature/journal/v431/n7011/full/nature03001.html.
- ^ Nystedt, Björn; Street, Nathaniel R.; Wetterbom, Anna. ”The Norway spruce genome sequence and conifer genome evolution” (på engelska). Nature 497 (7451): sid. 579-584. doi:. ISSN 0028-0836. http://www.nature.com/nature/journal/v497/n7451/full/nature12211.html.
- ^ Consortium, The Potato Genome Sequencing. ”Genome sequence and analysis of the tuber crop potato” (på engelska). Nature 475 (7355): sid. 189-195. doi:. ISSN 0028-0836. http://www.nature.com/nature/journal/v475/n7355/full/nature10158.html.
- ^ [a b] Chandrasekaran C & Betrán E. (2008). ”Origins of new genes and pseudogenes”. Nature Education 1: sid. 181.
- ^ [a b] Dillon, Niall. ”Gene regulation and large-scale chromatin organization in the nucleus” (på engelska). Chromosome Research 14 (1): sid. 117-126. doi:. ISSN 0967-3849. https://link.springer.com/article/10.1007/s10577-006-1027-8. Läst 8 januari 2016.
- ^ Hong, Joung-Woo; Hendrix, David A.; Levine, Michael S.. ”Shadow Enhancers as a Source of Evolutionary Novelty” (på engelska). Science 321 (5894): sid. 1314-1314. doi:. ISSN 0036-8075. http://www.sciencemag.org/content/321/5894/1314. Läst 8 januari 2016.
- ^ Phillips P & Hoopes L. (2008). ”Transcription factors and transcriptional control in eukaryotic cells”. Nature Education 1: sid. 118.
- ^ Matthews, B. W.; Ohlendorf, D. H.; Anderson, W. F.. ”Structure of the DNA-binding region of lac repressor inferred from its homology with cro repressor” (på engelska). Proceedings of the National Academy of Sciences 79 (5): sid. 1428-1432. ISSN 0027-8424. http://www.pnas.org/content/79/5/1428. Läst 8 januari 2016.
- ^ Wintjens, René; Rooman, Marianne. ”Structural Classification of HTH DNA-binding Domains and Protein – DNA Interaction Modes”. Journal of Molecular Biology 262 (2): sid. 294-313. doi:. http://www.sciencedirect.com/science/article/pii/S0022283696905148. Läst 8 januari 2016.
- ^ Klug, A.; Rhodes, D.. ”Zinc Fingers: A Novel Protein Fold for Nucleic Acid Recognition” (på engelska). Cold Spring Harbor Symposia on Quantitative Biology 52: sid. 473-482. doi:. ISSN 0091-7451. http://symposium.cshlp.org/content/52/473. Läst 8 januari 2016.
- ^ Klug, Aaron. ”Zinc finger peptides for the regulation of gene expression”. Journal of Molecular Biology 293 (2): sid. 215-218. doi:. http://www.sciencedirect.com/science/article/pii/S0022283699930073. Läst 8 januari 2016.
- ^ [a b] Krishna, S. Sri; Majumdar, Indraneel; Grishin, Nick V.. ”Structural classification of zinc fingers SURVEY AND SUMMARY” (på engelska). Nucleic Acids Research 31 (2): sid. 532-550. doi:. ISSN 0305-1048. http://nar.oxfordjournals.org/content/31/2/532. Läst 8 januari 2016.
- ^ [a b] Chaudhary, Jaideep; Skinner, Michael K.. ”Basic Helix-Loop-Helix Proteins Can Act at the E-Box within the Serum Response Element of the c-fos Promoter to Influence Hormone-Induced Promoter Activation in Sertoli Cells”. Molecular Endocrinology 13 (5): sid. 774-786. doi:. ISSN 0888-8809. https://academic.oup.com/mend/article/13/5/774/2741755. Läst 8 januari 2016.
- ^ [a b] Landschulz, W.; Johnson, P.; McKnight, S.. ”The leucine zipper: a hypothetical structure common to a new class of DNA binding proteins” (på engelska). Science 240 (4860): sid. 1759-1764. doi:. http://dx.doi.org/10.1126%2fscience.3289117. Läst 8 januari 2016.
- ^ [a b c] Vinson, C. R.; Sigler, P. B.; McKnight, S. L.. ”Scissors-grip model for DNA recognition by a family of leucine zipper proteins” (på engelska). Science 246 (4932): sid. 911-916. doi:. ISSN 0036-8075. http://www.sciencemag.org/content/246/4932/911. Läst 8 januari 2016.
- ^ Thomas, J. O.. ”HMG1 and 2: architectural DNA-binding proteins” (på engelska). Biochemical Society Transactions 29 (4): sid. 395-401. doi:. ISSN 0300-5127. http://www.biochemsoctrans.org/content/29/4/395. Läst 8 januari 2016.
- ^ Štros, M.; Launholt, D.; Grasser, K. D.. ”The HMG-box: a versatile protein domain occurring in a wide variety of DNA-binding proteins” (på engelska). Cellular and Molecular Life Sciences 64 (19-20): sid. 2590-2606. doi:. ISSN 1420-682X. https://link.springer.com/article/10.1007/s00018-007-7162-3. Läst 8 januari 2016.
- ^ Yamasaki, Kazuhiko; Kigawa, Takanori; Inoue, Makoto. ”Solution Structure of the B3 DNA Binding Domain of the Arabidopsis Cold-Responsive Transcription Factor RAV1” (på engelska). The Plant Cell 16 (12): sid. 3448-3459. doi:. ISSN 1532-298X. http://www.plantcell.org/content/16/12/3448. Läst 8 januari 2016.
- ^ Lohse, Matthew B.; Hernday, Aaron D.; Fordyce, Polly M.. ”Identification and characterization of a previously undescribed family of sequence-specific DNA-binding domains” (på engelska). Proceedings of the National Academy of Sciences 110 (19): sid. 7660-7665. doi:. ISSN 0027-8424. http://www.pnas.org/content/110/19/7660. Läst 8 januari 2016.
- ^ [a b] Bürglin, Thomas R.; Affolter, Markus. ”Homeodomain proteins: an update” (på engelska). Chromosoma: sid. 1-25. doi:. ISSN 0009-5915. https://link.springer.com/article/10.1007/s00412-015-0543-8. Läst 8 januari 2016.
- ^ ”Nucleic Acid Recognition by OB-Fold Proteins”. Annual Review of Biophysics and Biomolecular Structure 32 (1): sid. 115-133. doi:. http://dx.doi.org/10.1146/annurev.biophys.32.110601.142506. Läst 8 januari 2016.
- ^ Teichmann, Martin; Dumay-Odelot, Hélène; Fribourg, Sébastien. ”Structural and functional aspects of winged-helix domains at the core of transcription initiation complexes”. Transcription 3 (1): sid. 2-7. doi:. ISSN 2154-1264. http://dx.doi.org/10.4161/trns.3.1.18917. Läst 8 januari 2016.
- ^ Weigel, Nancy L.; Moore, Nicole L.. ”Steroid Receptor Phosphorylation: A Key Modulator of Multiple Receptor Functions”. Molecular Endocrinology 21 (10): sid. 2311-2319. doi:. ISSN 0888-8809. https://academic.oup.com/mend/article/21/10/2311/2738226. Läst 8 januari 2016.
- ^ Brivanlou, Ali H.; Darnell, James E.. ”Signal Transduction and the Control of Gene Expression” (på engelska). Science 295 (5556): sid. 813-818. doi:. ISSN 0036-8075. http://www.sciencemag.org/content/295/5556/813. Läst 8 januari 2016.
- ^ Matys, V.; Kel-Margoulis, O. V.; Fricke, E.. ”TRANSFAC® and its module TRANSCompel®: transcriptional gene regulation in eukaryotes” (på engelska). Nucleic Acids Research 34 (suppl 1): sid. D108-D110. doi:. ISSN 0305-1048. http://nar.oxfordjournals.org/content/34/suppl_1/D108. Läst 8 januari 2016.
- ^ ”Gene Regulation”. www.gene-regulation.com. http://www.gene-regulation.com/pub/databases/transfac/cl.html. Läst 8 januari 2016.
Media som används på denna webbplats
(c) Zephyris from en.wikipedia.org, CC BY-SA 3.0
By Richard Wheeler (Zephyris) 2007.
Lambda repressor protein bound to a lambda operator DNA sequence. From .Författare/Upphovsman: Yikrazuul, Licens: CC BY-SA 3.0
leucine zipper: CREB-1 binding to DNA. Data used from 1dh3. The magnesium ion is colored in green